物理化学と計算科学の手法を併用し
タンパク質の立体構造を解析する
生物が持っている膨大で複雑な情報をコンピュータで解析することが可能になった現代、生物学と情報科学を融合した「バイオインフォマティクス(生物情報科学)」が急速に発展している。小島正樹教授は、従来の物理化学の手法とともにバイオインフォマティクスや計算科学を駆使し、タンパク質をはじめとした生体の分子構造を論理的に解明し、in silico(コンピュータ上)で病態解明や新薬創製につなげようとしている。
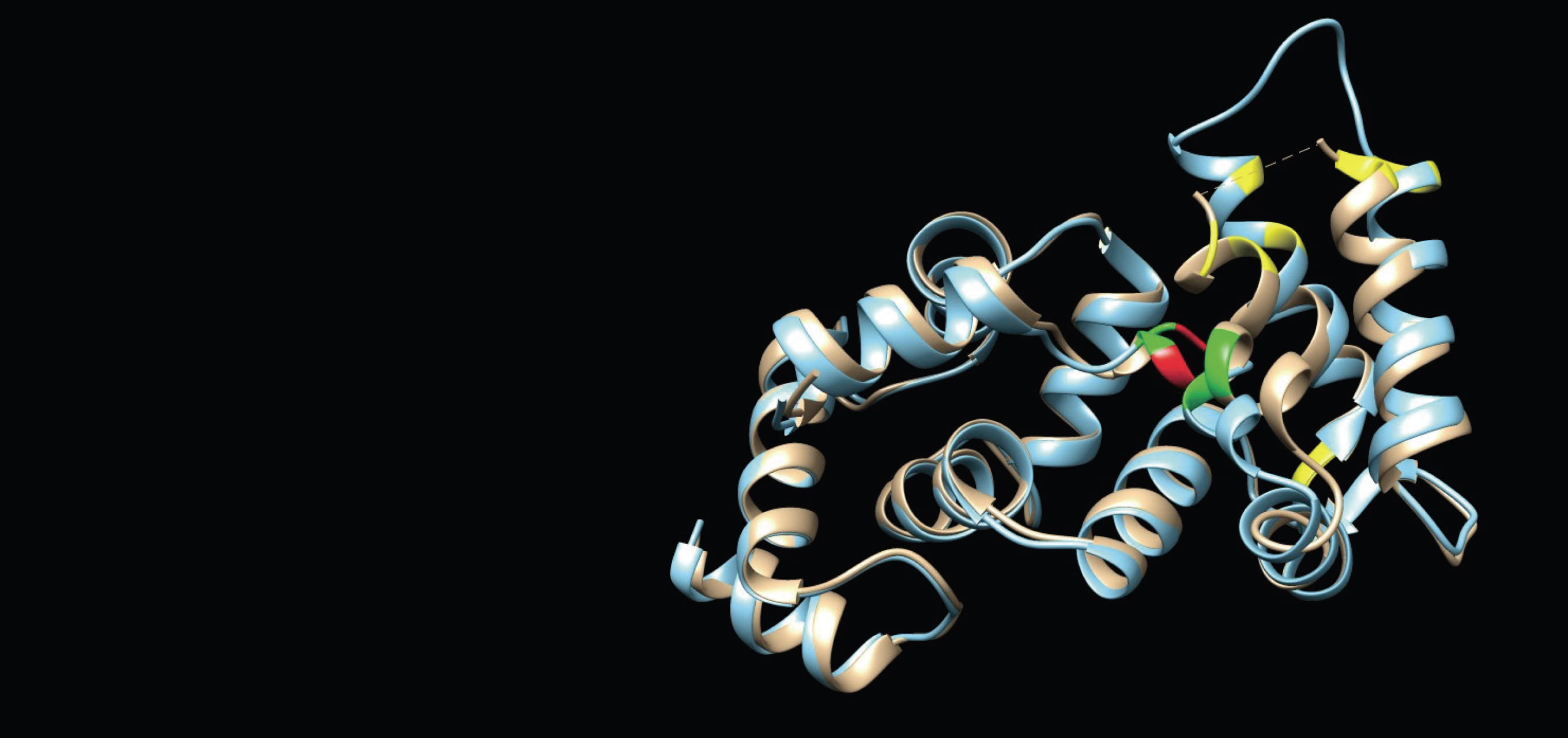
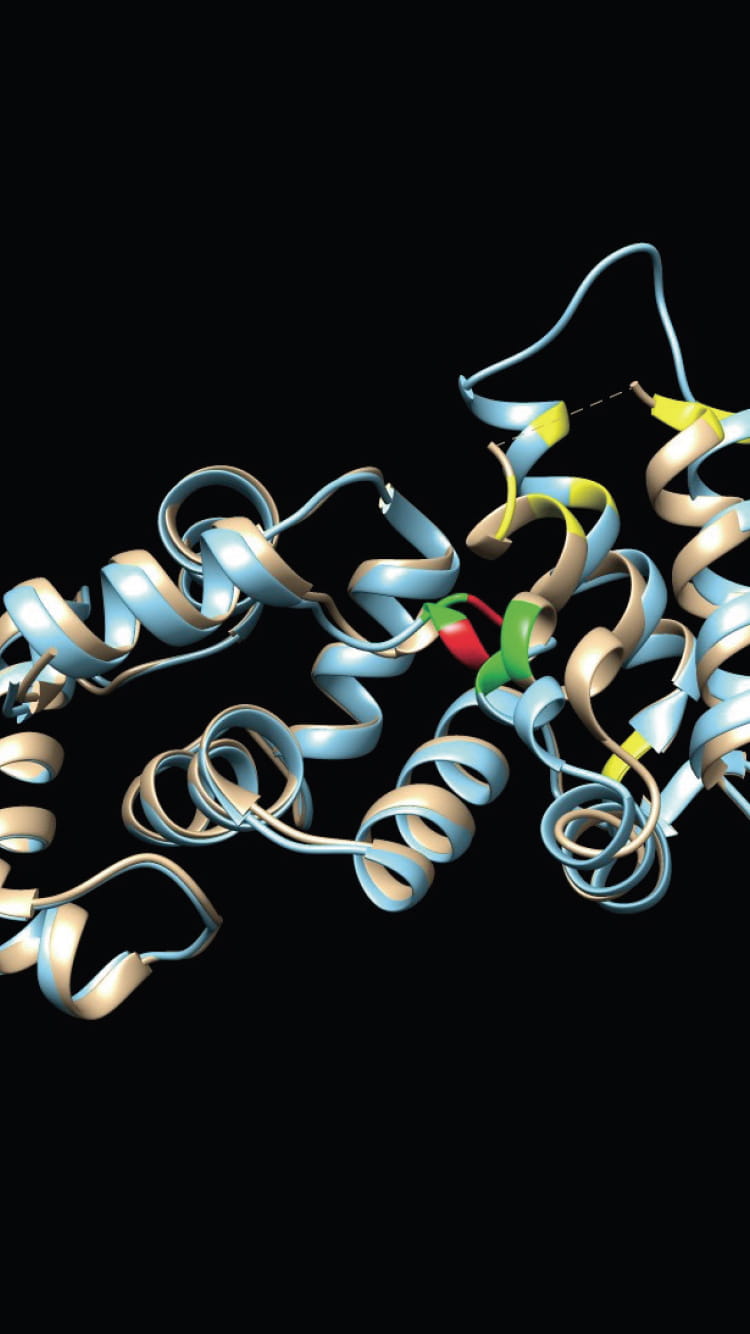
研究の柱の一つが、疾患に関わるタンパク質の立体構造を解析することである。タンパク質の構造解析には、一般にX線結晶解析やNMR(核磁気共鳴)法、電子顕微鏡による観察などさまざまな手法があるが、小島教授は物理化学の解析手法に計算科学の手法を組み合わせた新しい解析法を開発し、従来の方法では解析の難しかったタンパク質の立体構造の解明を可能にした。
小島教授が物理化学的手法として活用するのが、大学共同利用機関法人高エネルギー加速器研究機構の有する「X線溶液散乱(SAXS)」だ。「SAXSは試料にX線を照射し、散乱するX線強度の角度分布から構造情報を得る手法です。立体構造のモデルを与えると、散乱理論に基づいて散乱データを正確に計算できるのというメリットがあります」と小島教授。教授は、実測した散乱強度とモデルから計算した散乱強度のずれを束縛条件とし、束縛条件下での各原子の動きから実測データを満たす立体構造を導き出すアルゴリズムを構築。SAXSとこの「束縛条件付きMD(分子動力学)法」を併用し、迅速かつ簡便にタンパク質の立体構造を解析する方法を開発した。
現在は国内屈指の大学や研究機関からも依頼され、タンパク質の解析を請負っている。「タンパク質の機能は、アミノ酸のフォールディングによってできる立体構造によって決定されます。そのため立体構造を解明して初めて病気の原因分子を突き止め、治療法を見出すことも可能になります」と構造解析の意義を語る小島教授。アガリクスというキノコに含まれるβ-グルカンの立体構造を明らかにしたのも成果の一つだ。β-グルカンには抗腫瘍活性があることが知られている。立体構造の解明が、新たな抗がん剤の開発につながるかもしれない。
医薬品候補の分子構造をコンピュータで設計する
研究の二つ目の柱が、「in silico創薬」である。高速の汎用計算機(GPGPUワークステーション)を使い、MDシミュレーションなどの計算手法によって、有用な医薬品候補分子の構造を設計するという。最近大きなインパクトを与えたのが、新型コロナウイルス感染症(COVID-19)に対する新規治療薬の創製につながる研究である。
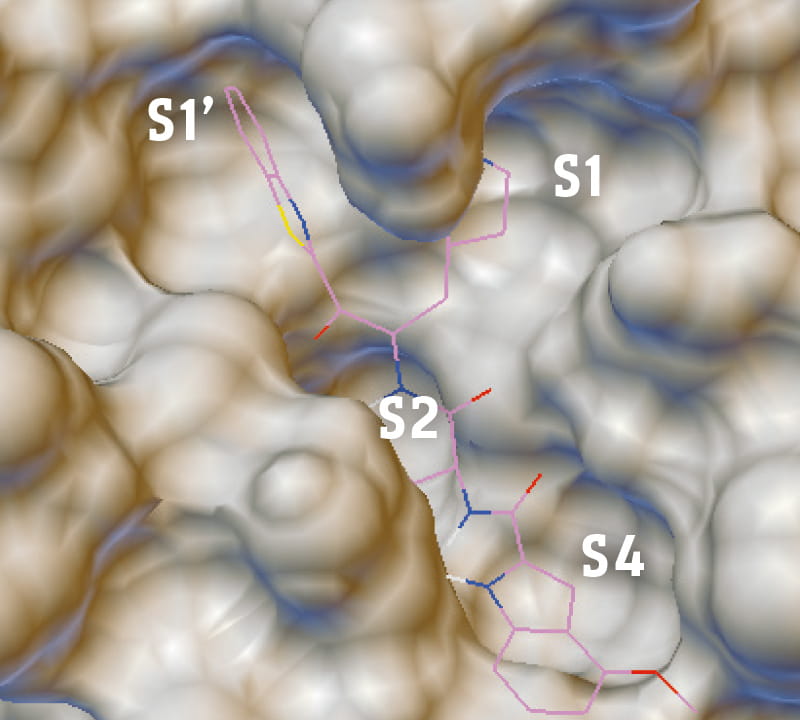
「共同研究を行う本学薬学部の林 良雄教授は、2002年に流行したSARS(重症急性呼吸器症候群)の原因ウイルスSARS-CoVについて研究し、ウイルス増殖に関与する3CLプロテアーゼの働きを阻害するSARS-CoVのメインプロテアーゼ阻害剤YH-53を創製しています。COVID-19の原因ウイルスであるSARS-CoV-2は、SARS-CoVと極めて高い相同性があることが報告されており、私たちはYH-53がSARS-CoV-2にも効果を発揮するのではないかと考えました」と小島教授は研究の発端を語る。
しかし薬効が見つかったからといってすぐに薬を作れるわけではない。実際にヒトの体内で効力を発揮する化合物にし、一方で害を与える毒性を排除するなど、創薬までには多くのハードルを乗り越える必要がある。小島教授の研究グループは、ドッキングシミュレーションを用いてSARS-CoV-2プロテアーゼ阻害剤として最適化するべくin silicoで分子構造を設計。さらにケモインフォマティクスと機械学習の手法を用いて細胞毒性や薬物動態を予測し、データサイエンスの視点から医薬品としての適性を評価・確認した。
本研究は、国立研究開発法人日本医療研究開発機構(AMED)の事業に採択され、新型コロナウイルス感染症の治療薬開発につながると大きな期待を集めている。
「候補分子から新薬開発が成功する確率は、数万分の一、開発期間は平均15年以上に及ぶといわれています。有効成分を見つけることができても、『死の谷』といわれる臨床試験を乗り越え、上市まで至るのは、ほんの一握りです。in silicoで立体構造の理論的構築とビッグデータに基づく薬物動態・毒性特性の予測を行うことで、創薬の飛躍的な迅速化と高精度化が可能になります」と小島教授は言う。
未知の分子動態を予測する独自プログラムを開発
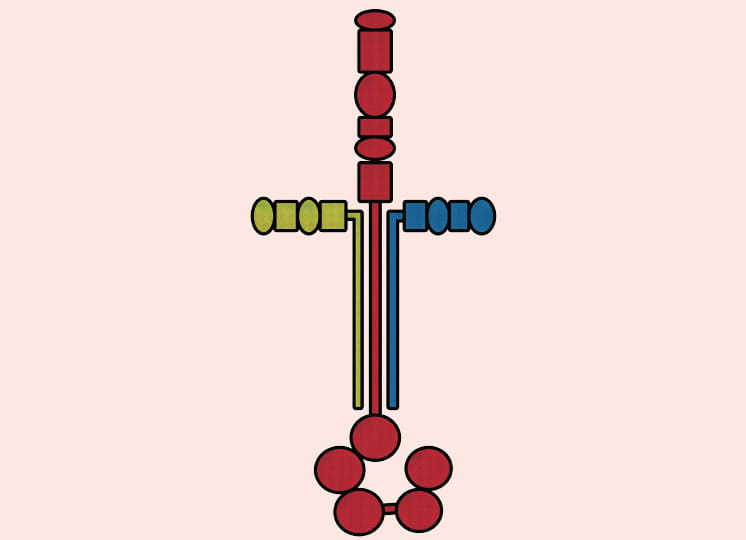
さらに三つ目の研究が、「VOLTES(Virtual Optimization of Local TErtiary Structures)」プロジェクトだ。「VOLTES」は、小島教授らが開発した独自プログラムのこと。「タンパク質の立体構造データベース『Protein Data Bank』に登録されているビッグデータを、バイオインフォマティクス、位相幾何学、機械学習の手法を使ってマイニングし、タンパク質の構造解析や構造変化予測を行います」と語る。
プログラムの特徴は、タンパク質の立体構造とトポロジー情報を二面角の「木」で表現するとともに、二面角の数値を六進数で表現するところにある。「VOLTES」で解析することで、実験では捉えることのできない分子の構造変化を迅速かつ正確に求めることが可能になる。「『VOLTES』でタンパク質の未知の構造を明らかにし、in silico創薬への応用を目指します」と未来を見据える。